瀬戸臨海実験所教育拠点研究員の佐藤 崇さんが,もう一本論文を出版されました.
おめでとうございます!
論文タイトルと概要は以下の通りです.
⬛︎ Structure and variation of the mitochondrial genome of fishes.
Satoh, T. P., M. Miya, K. Mabuchi, and M. Nishida.
BMC Genomics, 2016, 17:719.
DOI: 10.1186/s12864-016-3054-y
URL: https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-016-3054-y
概要
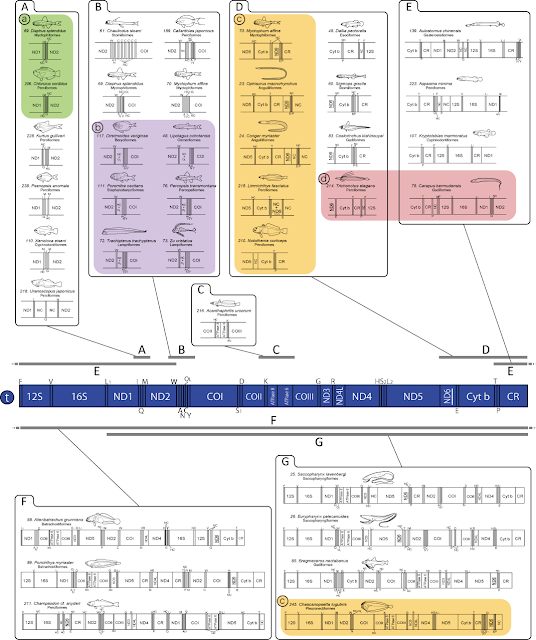
ミトコンドリア (mt) ゲノムは,これまで多くの脊椎動物の進化研究に用いられてきました.しかし,そこにコードされる遺伝子に関する共通構造や不変領域の位置などの基礎情報,そして近年報告が増えている遺伝子配置変動について,大量データに基づき比較した研究は多くありません.そこで,脊椎動物の中で最も mt ゲノム全塩基配列データが蓄積されている魚類に注目し,これらの大規模データを詳細に比較することにより,mt ゲノムの基礎構造から変異特性までを網羅的に捉え,魚類 mt ゲノムを特徴づけることを目的としました.
具体的には,条鰭類 248 種に軟骨魚類 2 種を加えた 250 種の mt ゲノムデータを元に,各遺伝子の比較解析を行いました.タンパク質遺伝子については,各遺伝子の開始・終止コドンの変異性や使用頻度,また遺伝子長や高次構造などを比較し,その特徴を整理しました.tRNA や rRNA 遺伝子に関しては,遺伝子長や保存的な配列などについて比較し,それらの結果を統合して,魚類における各遺伝子の代表的な二次構造モデルを決定しました.また,魚類 250 種の mt ゲノムから 32 科 35 種 (35/250 = 14%) で遺伝子配置変動例が見つかりました.さらにこれらの変動は,tRNA 遺伝子がクラスターを形成している IQM,WANCY 領域,そして ND5 から調節領域 (CR) にかけての領域に多く生じていることが明らかになりました.このような配置変動の多くは,科もしくは属間における有効な系統マーカーとなります.しかし,系統的に離れている種間で,部分的に同じ遺伝子配置を共有している例も判明しました.このことから,mt ゲノムの部分領域における遺伝子配置パターンの共有のみを,単系統性のマーカーとして単純に扱うことで解析結果に誤解を生じさせる原因となりうることが明らかになりました.
本研究のような,大量のデータを基盤にした詳細な比較研究により,初めて魚類mtゲノムの基本構造や共通領域などの基礎情報が明らかになり,それを元に構造的特徴やその違い,遺伝子配置変動の進化パターンなどが可視化することができました.このような情報は,近年の大規模塩基配列データをもちいた進化研究や遺伝子のアノテーションシステムの開発などに大きく貢献するだけではなく,mt ゲノム自体の進化を理解するための重要な情報を多数含んでいるのではないかと考えています.